PCR steht für die englische Bezeichnung Polymerase Chain Reaction, auf deutsch Polymerasekettenreaktion. Entwickelt wurde der Test 1983 von Kary Mullis. Er erhielt dafür 1993 den Nobelpreis für Chemie. Die Entwicklung war ein Meilenstein für die molekularbiologische Forschung.
Grundlegende Funktionsweise
Der PCR-Test ist eine Art Vervielfältiger für DNA-Sequenzen außerhalb des lebenden Organismus. Dafür nutzt man Enzyme und Bausteine, die auch in den Körperzellen für die Verdopplung der DNA zuständig sind. Ein Verfielfältigungszyklus wird als Threshold Cycles (CT-Wert) bezeichnet.
Die Ausgangs DNA (Adenin, Guanin, Thymin und Cytosin) wird mit der DNA-Polymerase, ein Enzym das die DNA-Bausteine zusammensetzen kann, und den genspezifischen Primern, die der Polymerase zeigt wo sie mit der Zusammensetzung beginnen soll, in ein Reaktionsgefäß gegeben.
Ablauf eines Threshold Cycles (CT-Wert)
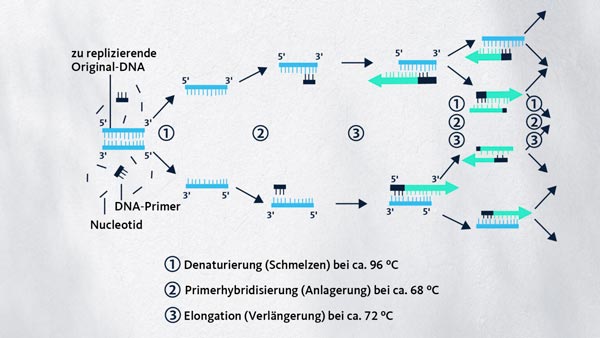
Denaturierung
Das Reaktionsgefäß wird in einem Thermocycler auf 96 Grad Celsius erhitzt. Die Doppelhelix der DNA wird dadurch in zwei komplementäre Stränge aufgeteilt.
Anlagerung
Die Temperatur im Thermocycler wird auf 68 Grad Celsius gesenkt. Nun können die genspezifischen Primer ihre Arbeit aufnehmen. Wenn der Primer eine passende Gensequenz auf den komplementären Strängen findet kann er sich daran anlagern.
Verlängerung
Bei 72 Grad Celsius erolgt nun der Verlängerungsvorgang. Ausgehend von den Primern bauen die Polymerasen einen neuen Strang an die freiliegenden Stränge der Ausgangs-DNA an. Aus einer Ausgangs-Doppelstrang-DNA werden zwei. Der erste Threshold Cycle ist abgeschlossen. Danach wird wieder mit der Denaturierung begonnen.
Specific Enzymatic Amplification of DNA In Vitro: The Polymerase Chain Reaction
K. MULLIS, F. FALOONA, S. SCHARF, R. SAIKI, G. HORN, AND H. ERLICH
Cetus Corporation, Department of Human Genetics, Emeryville, California 94608




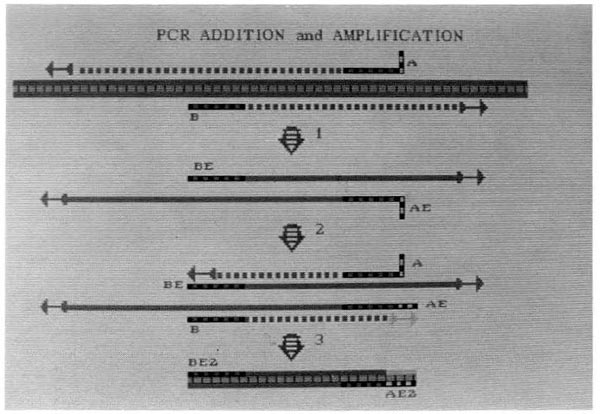
Abbildungen A, B, C und D. Oben sind drei vollständige Zyklen der Polymerase-Kettenreaktion dargestellt, die zur achtfachen Vermehrung einer Vorlagesequenz führen, die durch die 5′-Enden von zwei Primern definiert ist, die an verschiedene Stränge der Vorlage hybridisiert sind. Der erste Zyklus ist im Detail als Reaktion 1, 2 und 3 dargestellt. Der zweite Zyklus ist weniger detailliert als Reaktionen 4 und 5 und der dritte Zyklus als Reaktionen 6 und 7 dargestellt. In Zyklus 3 wird ein doppelsträngiges DNA-Fragment hergestellt, das diskrete Termini aufweist, und somit wurde die Zielsequenz sowohl aus der Quelle extrahiert als auch amplifiziert.

Eine PCR-Amplifikation mit den Oligonukleotiden A und B ergibt nach einer Anzahl von Zyklen das Fragment AE: BE. Ein Teilfragment, CE:DE, kann aus diesem Produkt extrahiert und mit Hilfe der Oligonukleotide C und D weiter amplifiziert werden. Dieser Gesamtprozess erfordert vier unabhängige und koordinierte Primer-Vorlage-Interaktionen und ist damit wesentlich spezifischer als eine einstufige PCR-Amplifikation.
Die Addition der nichthomologenen Sequenz am 5′-Ende des Oligonukleotids A führt nach dem ersten PCR-Zyklus zum Einbau dieser nichthomologenen Sequenz in das Verlängerungsprodukt AE, das in einem späteren Zyklus als Vorlage für B die Synthese von BE2 steuert, und das zusammen mit AE2 das doppelsträngige DNA-Fragment AE2: BE2 bildet. Dieses Fragment wird bei weiteren Zyklen exponentiell vermehrt.
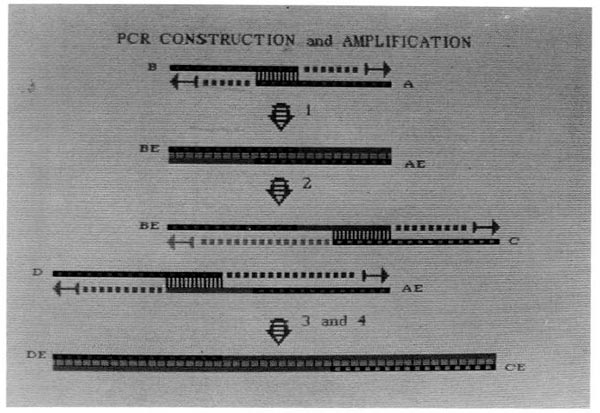
PCR-Konstruktion und Amplifikation. Oligonukleotide mit komplementären, überlappenden 3′-Enden werden mit Hilfe der DNA-Polymerase aneinander verlängert. Das doppelsträngige DNA-Produkt dieser Reaktion wird durch eine zweite Stufe von mehreren PCR-Zyklen dargestellt und durch die Reaktionen 2, 3 und 4, an beiden Enden verlängert.
Allerdings zerren die ständigen Temperaturwechsel an der Polymerase und den Primern. Eine PCR kann deshalb nicht endlos laufen. Im Falle der Corona-Tests ist meistens nach 40 bis 42 Zyklen Schluss. Ein hoher CT-Wert steht für eine geringe Menge an genomspezifischen Nukleotiden im Ausgangsmaterial.
Sensitivität und Spezifität
Kein Test ist fehlerfrei. Positive und negative Testergebnisse können also falsch liegen. Wie verlässlich ein bestimmtes Testverfahren ist, wird durch zwei Parameter bestimmt:
- Die Sensitivität eines Tests gibt an, bei wie viel Prozent der Proben die genomspezifischen Nukleotide auch wirklich erkannt werden. Ein Test mit einer Sensitivität von 99 Prozent identifiziert 99 von 100 Proben als positiv und eine nicht. Eine Person hat also ein falsch-negatives Ergebnis. Heißt: Je höher die Sensitivität ist, desto sicherer erfasst ein Test das Vorhandensein von genomspezifischen Nukleotiden.
- Die Spezifität gibt an, zu wie viel Prozent ein Test falsch positive Ergebnisse liefert. Ein Test mit einer Spezifität von 95 Prozent liefert bei 5 von 100 Probanden ein falsch positives Ergebnis. Genomspezifische Nukleotide sind nur in sehr geringen Mengen oder gar nicht vorhanden.
| Mögliche Testausgänge | ||
|---|---|---|
| Nukleotide | Keine Nukleotide | |
| Test positiv | Positiv (P) | Falsch positiv (FP) |
| Test negativ | Falsch negativ (FN) | Negativ (N) |
Sensitivität = P / (P + FN) und Spezifität = N / (N + FP)
Bedeutet ein positiver Test, dass man erkrankt ist?
Nein, der Test weist auf keine Erkrankung hin, sondern nur, dass man spezifische Nukleotide in sich trägt. Ob man diese weitergeben kann, hängt auch davon ab, wie hoch die Kontentration im Körper ist. Die Menge der genomspezifischen Nukleotide kann man über den CT-Wert abschätzen.
Fazit
Testergebnisse müssen im Kontext interpretieren werden. Massentests machen keinen Sinn. Ab einem CT-Wert von 25 liegt die Zuverlässigkeit des Tests bei ca. 70%, bei einem CT-Wert von 30 bei 20% und ab 35 Zyklen nurmehr bei 3%. Freiland Magazin berichtete darüber.

Foto: idexx.de